인리치먼트 분석을 위해 맞춤형 BED 파일을 매니페스트 파일로 변환하는 방법
03/23/22
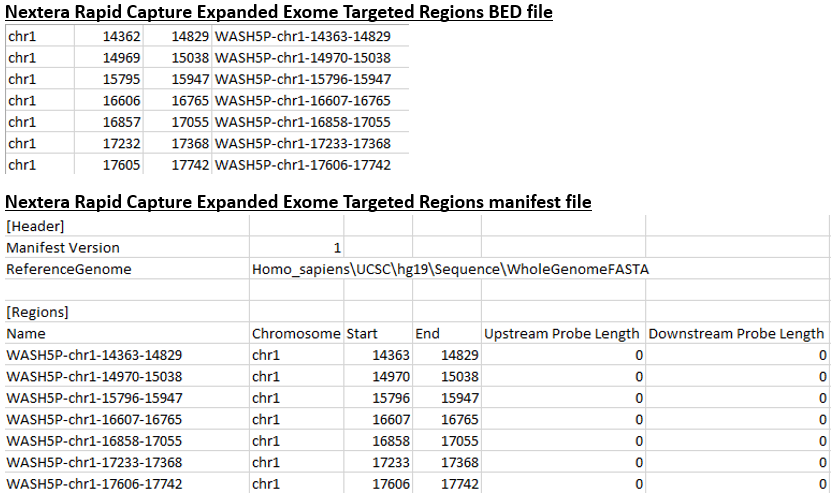
BaseSpace Sequence Hub , Local Run Manager 및 MiSeq Reporter 의 Illumina Illumina Enrichment 분석 워크플로우를 포함하여 사용 가능한 Illumina 인리치먼트 분석 워크플로우는 Illumina 고정 패널 매니페스트를 사용하여 변이 검출을 위한 표적 영역을 지정하거나 맞춤형 매니페스트 파일을 사용할 수 있습니다. Illumina DesignStudio를 통해 생성된 Illumina 맞춤형 패널에 대한 적하목록이 제공됩니다. 또는, 많은 비Illumina 벤더가 적하목록으로 변환해야 하는 표적 영역을 정의하기 위해 BED 파일을 제공합니다. 아래 이미지와 같이 BED 파일(상단)과 매니페스트(하단)의 콘텐츠는 유사하지만 매니페스트 파일의 형식과 추가 정보가 다릅니다. 이 예제는 Nextera Rapid Capture Expanded Exome 콘텐츠 세트에서 가져온 것입니다.
BED 파일을 인리치먼트 매니페스트 파일로 변환하려면 다음 단계를 수행합니다.
- Nextera Rapid Capture Expanded Exome manifest를 다운로드하여 템플릿으로 사용하세요. 참조 유전체 경로를 포함한 [헤더] 섹션을 보관합니다.
- [Regions] 섹션의 경우 BED 파일의 열 4에서 적하목록 파일의 이름 열(열 1)로 표적 이름을 복사합니다. 자세한 지침은 아래 '중요 고려사항'을 참조하세요.
- BED 파일의 첫 번째, 두 번째 및 세 번째 열을 매니페스트 파일의 두 번째, 세 번째 및 네 번째 열(즉, 염색체, 시작, 종료)로 각각 복사합니다.
- 벤더가 업스트림 및 다운스트림 프로브 길이를 제공하는 경우, 다음 2개 열에 포함시킬 수 있습니다. 제공되지 않는 경우, 이 값을 0으로 설정합니다.
- CSV 또는 Excel이 아닌 탭 구분 텍스트 파일(TSV)로 저장합니다.
중요한 고려사항
- 표적 이름은 고유해야 합니다. 이름이 고유한지 확인하기 위해 Target.001, Target.002, Target.003과 같은 일련의 숫자 값을 추가할 수 있습니다.
- 표적 이름에는 문자, 숫자, 마침표, 대시 및 밑줄만 허용됩니다. 공백 및 특수 문자(예: : 및 ')는 허용되지 않습니다. < > 기호에 포함된 트랙 정보가 포함된 줄을 모두 제거합니다.
- BaseSpace Sequence Hub 로 가져올 때 고유하지 않은 표적 이름 또는 지원되지 않는 문자의 사용은 오류 메시지 “[Probes] 섹션이 누락되었거나 유효하지 않습니다”를 반환합니다. 인리치먼트 매니페스트 파일에는 [Probes] 섹션이 필요하지 않지만 이 메시지는 파일 형식이 유효하지 않음을 나타냅니다.
- 매니페스트 파일을 BaseSpace Sequence Hub 가져올 때 크기가 30MB를 초과하는 파일은 검증 단계에 실패할 수 있습니다.